ITS2 RefSeq Integration Workflow(IRW)
本ワークフローは、環境DNA分析に用いるDNAデータベースの構築、生物種をアノテーションする分類器の構築、複数の分類器を使用した生物種アノテーション、およびその複数の結果を統合する一連のフローである。使用する分類器はQiime2で提供されているナイーブベイズ分類器、Blastの2種類である。本ワークフローを構成する3つの部分は基本的に独立に機能するため、使用したい工程のみを利用することも可能である。
1. はじめに
NCBIに収録されているDNAデータは、基本的に登録に必要な条件さえ満たしていれば収載可能なデータであり、その登録内容が精査されているわけではない。そのため、キュレーションを行った様々なデータベースが発表されており、植物ITS2領域に関しても複数のデータベースが存在する。しかし、既存のデータベースはどのようなキュレーションをおこなったのか、詳細までは公表されておらずデータベースの精度を確認する作業などが必要である。また、公開から時間が経過し未更新であるものも存在する。そこで、データベースの構築手順についてDuboisらにより提唱されたUnixベースの参照用データベース作成フローを用いてユーザーが自身で最初から構築を行える手順を作成した。なお、pythonを利用することでより簡便に構築が進められる部分等、ワークフローに一部変更を加えている。(ITS2 RefSeq Integration Workflow-1)
本ワークフローでは分類器の使用に関してもモデル事例を作成している(ITS2 RefSeq Integration Workflow-2)。
植物ITS2領域の分析例として多いものにハチミツの中の花粉DNA分析がある。Qiime2のfeature-classifyプラグインの特徴は、あるレベルの生物分類に複数の同程度にもっともらしい生物種が該当した場合、当該分類レベルでの判別を行わないため、結果として上位分類レベルに判別が止まる。しかしながら、実際に解析を行ってみるとハチミツが採取された地域に自生していない、混入が考えられない植物が種レベルで特定される例が発生する。また、上位分類の同定に配列が止まり、ハチミツDNA分析の目的である「種レベルでの同定」ができず、配列数で見たときに大きい割合で種レベルに特定できない解析例が確認された。前者は、ASV配列毎に判定結果に対する信頼性が異なっていることを考慮していないことが原因であるが、大量のASV配列を手作業で再確認していくのは非常に労力を有する。
そこでBlast検索の結果をQiime2の結果を照らし合わせ、統合的に判定できるワークフローを作成した。(ITS2 RefSeq Integration Workflow-3)。IRWの一連のワークフローにより、従来の判別方法(公表されている既存データベースを用いた単一分類器での判別)と比べて、種レベルでの同定割合と、誤判定が大きく改善した。例えば、従来の判別方法では、ハチミツの主要蜜源であるゲンゲ属レンゲをゲンゲ属ナンコオギ(台湾の一部に自生する希少なゲンゲ属)と判定していたが、レンゲ属ゲンゲに正しくアノテーションされるようになっている。
Dubois, B., Debode, F., Hautier, L., Hulin, J., San Martin, G., Delvaux, A., Janssen, E., & Mingeot, D. (2022). A detailed workflow to develop QIIME2-formatted reference databases for taxonomic analysis of DNA metabarcoding data. BMC Genetics, 23, Article 67. https://doi.org/10.1186/s12863-022-01067-5 (PMID: 35804326, PMCID: PMC9264521)
2. 内容
IRWは3つの部分で構成されている。
- ITS2 RefSeq Integration Workflow-1
- NCBI等から得たDNAデータをキュレーションし高品質なデータベースを構築する。加えて生物種をアノテーションするための分類器を構築する。
- Qiime2用のナイーブべイズ分類器、Blast検索用データベースの2種類を作成
- ITS2 RefSeq Integration Workflow-2
- 作成した分類器を用いて生物種をアノテーションするプロセスである。本ワークフローではTrimmaticを用いるため事前のインストールが必要である。
- ITS2 RefSeq Integration Workflow-3
- Qiime2 での生物分類結果と、Blast検索での生物分類結果を統合する工程である。
3.特徴
本フローにより従来の判定例から種レベルの判定実施率と同時に結果の信頼性を大幅に向上させることが可能である。
以下は、愛知県内で採取されたハチミツ5サンプルを分析し得られた結果を評価したものである。
PLANiTs:PLANiTsで提唱されたデータベースを用い、Qiime2feature-classiferで判別を行ったケース
IRW-1:ITS2 RefSeq Integration Workflow-1により作成されたデータベースを用いてQiime2feature-classiferで判別を行うケース
Blast_best_match: IRW-1により作成されたBlast検索用データベースを用いBlast検索し判別を行うケース
IRW-3:ITS2 RefSeq Integration Workflow-1~3全ての工程を用いて判別を行ったケース
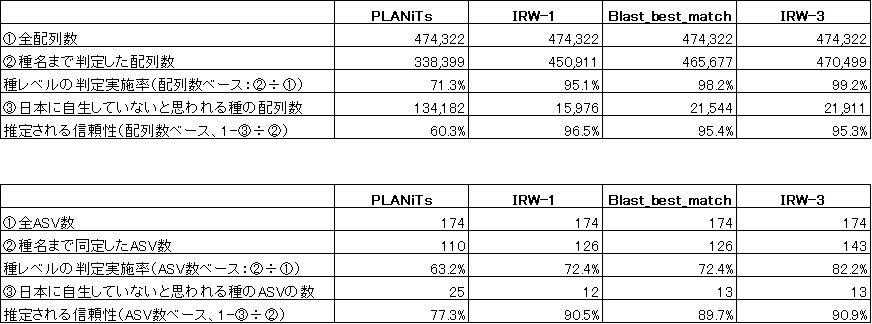
*日本に自生していない種はGBIFウェブサイト等で確認
*PLANiTs:Banchi, E., Ametrano, C. G., Greco, S., Stanković, D., Muggia, L., & Pallavicini, A. (2020). PLANiTS: A curated sequence reference dataset for plant ITS DNA metabarcoding. Database, 2020, baz155. https://doi.org/10.1093/database/baz155
*日本に自生していないと思われる種であっても、Qiime2 feature classiferおよびBlast検索の結果は非常に高いスコアに該当しているものが複数あり、データ解析の結果としては問題ないものも含まれる。
4.構築環境
使用マシン:Microsoft Azure 仮想マシン Standard E16-8ads v5 (8 vcpu 数、128 GiB メモリ)
オペレーティングシステム:Linux
使用バージョン:Qiime2_2023_07
5.留意事項
導入をご検討されるお客様は、弊社分析部 contact@bioinsight.co.jp までご連絡下さい。その他、ご質問やご感想、ご意見等お待ちしております。
本ワークフローのご利用はユーザー様ご自身のご判断でお願いしております。本ワークフローにより被った損害等については弊社はその責を負いません。